Desbloqueando insights importantes sobre a expressão genética usando um novo modelo de camundongo

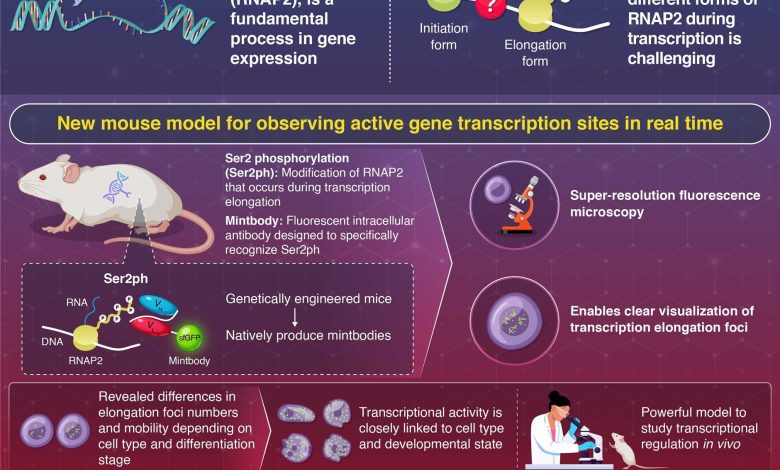
Este infográfico descreve a abordagem proposta para visualizar processos de transcrição in vivo. Crédito: Instituto de Ciência de Tóquio
Um modelo de rato recentemente desenvolvido permite a visualização em tempo real da RNA Polimerase II (RNAP2) durante a transcrição do DNA, conforme relatado por pesquisadores da Science Tokyo. A equipe projetou camundongos para produzir um anticorpo fluorescente que se liga especificamente ao RNAP2 durante a transcrição ativa. Sua abordagem permitiu imagens de super-resolução de locais de transcrição em células vivas, revelando diferenças na dinâmica de expressão gênica entre tipos de células e estados de desenvolvimento.
A transcrição é o processo celular pelo qual o RNA é produzido a partir do DNA – uma etapa fundamental na expressão gênica e, em última análise, na síntese de proteínas e moléculas funcionais de RNA. Um ator essencial na transcrição é a RNA Polimerase II (RNAP2), um grande complexo proteico que desenrola o DNA e coordena a síntese da cadeia de RNA.
A jornada do RNAP2 pode ser dividida em duas fases principais: iniciação e alongamento. Durante a iniciação, o complexo se instala no ponto inicial do gene e começa a construir o primeiro trecho curto de RNA. Após uma pausa, o RNAP2 muda para o alongamento, durante o qual ele viaja ao longo do gene enquanto estende continuamente a cadeia de RNA até que a transcrição completa seja produzida.
Para investigar detalhadamente como a expressão genética é controlada, os cientistas precisam observar o RNAP2 em ação. No entanto, as ferramentas atualmente disponíveis, como o RNAP2 marcado com fluorescência, não conseguem distinguir claramente entre as formas que o RNAP2 assume durante a iniciação e o alongamento.
Neste contexto, uma equipe de pesquisa liderada pelo professor Hiroshi Kimura do Centro de Biologia Celular, Instituto de Pesquisa Integrada do Instituto de Ciência de Tóquio, no Japão, desenvolveu um modelo inovador de camundongo que permite a visualização in vivo de RNAP2 durante o alongamento. Publicado no Jornal de Biologia Molecular em 13 de agosto de 2025, este estudo marca um passo crítico em direção a uma compreensão mais completa da expressão genética e seus mecanismos regulatórios in vivo.
O modelo se concentra na detecção da fosforilação de Ser2 (Ser2ph), uma importante modificação molecular que o RNAP2 sofre e mantém durante o alongamento. Os pesquisadores modificaram ratos geneticamente modificados para expressar um anticorpo intracelular fluorescente específico para modificação (também chamado de “mintbody”) que se liga especificamente ao RNAP2 modificado por Ser2ph. Usando microscopia de fluorescência de super-resolução, a equipe conseguiu visualizar claramente focos de alongamento da transcrição nos núcleos de células vivas de vários tecidos.
“Nosso método revelou padrões dinâmicos de atividade de transcrição genética em tecidos de camundongos que antes só eram observáveis em amostras fixas”, diz Kimura.
Os pesquisadores analisaram então como o alongamento da transcrição variava dependendo do tipo de célula e do estado de desenvolvimento, observando diferenças marcantes no número e na mobilidade dos locais de transcrição. Por exemplo, as células imunes no baço exibiram padrões de transcrição únicos, e as células em proliferação geralmente exibiram mais focos de transcrição móveis em comparação com aquelas em células diferenciadas (maduras).
No geral, esses experimentos mostram como a abordagem proposta pode esclarecer o intrincado processo de transcrição. “O modelo de rato recentemente desenvolvido oferece uma ferramenta poderosa para estudar a regulação transcricional in vivo, com amplas implicações para a compreensão do desenvolvimento, diferenciação, respostas ambientais, envelhecimento e mecanismos de doenças a nível celular”, conclui Kimura. “Também é uma promessa para aplicações futuras no desenvolvimento terapêutico e na medicina de precisão.”
Mais informações:
Chihiro Matsuda et al, Organização e Dinâmica de Focos de Alongamento de Transcrição em Tecidos de Rato, Jornal de Biologia Molecular (2025). DOI: 10.1016/j.jmb.2025.169395
Fornecido pelo Instituto de Ciência de Tóquio
Citação: Desbloqueando insights importantes sobre a expressão genética usando um novo modelo de camundongo (2025, 11 de novembro) recuperado em 11 de novembro de 2025 em https://medicalxpress.com/news/2025-11-key-insights-gene-mouse.html
Este documento está sujeito a direitos autorais. Além de qualquer negociação justa para fins de estudo ou pesquisa privada, nenhuma parte pode ser reproduzida sem permissão por escrito. O conteúdo é fornecido apenas para fins informativos.