Telas CRISPR em todo o genoma identificam sensibilidade e resistência ao inibidor de PARP no câncer de próstata

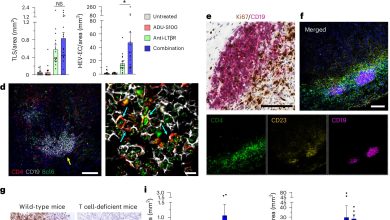
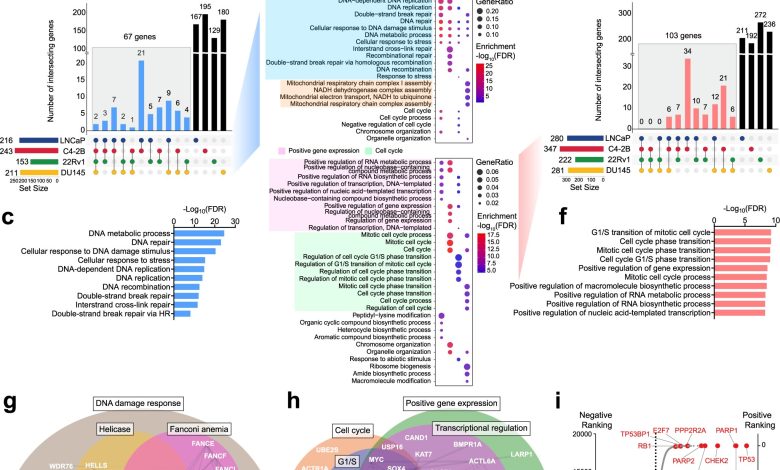
As telas CRISPR identificam genes que modulam a resposta PARPi em células PCa. um esquema de telas CRISPR/Cas9 em todo o genoma. b gráfico UpSet de genes selecionados negativamente em quatro linhagens de células PCa conforme indicado. As barras azuis indicam o número de acertos comuns em pelo menos duas telas. c Os principais termos GO enriquecidos em 67 ocorrências comuns de seleção negativa. d Termos GO superiores enriquecidos em genes selecionados negativamente (painel superior) e positivamente (painel inferior) em cada linha celular individual. e gráfico UpSet de genes selecionados positivamente em quatro linhagens de células PCa conforme indicado. As barras vermelhas indicam o número de acertos comuns em pelo menos duas telas. f Principais termos GO enriquecidos em 103 ocorrências comuns de seleção positiva. g As redes de acertos comuns da seleção negativa agrupadas de acordo com seus papéis em vias específicas e suas interações genéticas e físicas (linhas cinzas) com base na análise STRING. h As redes de acertos comuns da seleção positiva, agrupadas como em (g). i Os genes mais bem classificados das telas CRISPR determinados pela comparação do tratamento com olaparibe e DMSO. Os genes são classificados pela média de pontuações β diferenciais de todas as quatro linhagens de células. Os genes selecionados negativa e positivamente são marcados em azul e vermelho, respectivamente. Crédito: Natureza Comunicações (2023). DOI: 10.1038/s41467-023-35880-y
Os tumores de câncer de próstata que abrigam mutações BRCA1/2 são excepcionalmente sensíveis aos inibidores de PARP, enquanto alterações genômicas em outros genes de resposta a danos no DNA (DDR) são menos responsivos. Para identificar genes previamente desconhecidos cuja perda tem um impacto profundo na resposta do inibidor de PARP, pesquisadores do Dana-Farber Brigham Cancer Center lideraram um esforço multinacional para realizar telas de nocaute de CRISPR-Cas9 em todo o genoma. O objetivo do estudo foi informar o uso de inibidores de PARP além de tumores deficientes em BRCA1/2 e apoiar a reavaliação dos biomarcadores atuais para inibição de PARP no câncer de próstata.
As conclusões são publicadas na revista Natureza Comunicações.
O estudo identificou vários novos genes (por exemplo, MMS22L e RNASEH2B) que são frequentemente deletados em câncer de próstata. Esses genes podem servir como biomarcadores preditivos para a resposta do inibidor de PARP no câncer de próstata, de acordo com o estudo. A equipe de pesquisa também descobriu que a perda de CHEK2 (o biomarcador aprovado pela FDA para resposta terapêutica ao olaparibe) confere resistência, em vez de sensibilidade, à inibição de PARP.
“As atuais terapias direcionadas ao câncer, incluindo inibidores de PARP, são em grande parte guiadas por mutações de um único gene e negligenciam alterações genômicas concomitantes”, disse Li Jia, Ph.D., diretor de Pesquisa em Urologia do Brigham and Women’s Hospital. “Descobrimos que a sensibilidade do inibidor de PARP pode depender da interação entre múltiplas alterações genômicas. Portanto, uma análise genômica abrangente pode ajudar a melhorar a tomada de decisões clínicas.”
Mais Informações:
Takuya Tsujino et al, as telas CRISPR revelam determinantes genéticos da sensibilidade e resistência do inibidor de PARP no câncer de próstata, Natureza Comunicações (2023). DOI: 10.1038/s41467-023-35880-y
Fornecido por
Brigham and Women’s Hospital
Citação: Telas CRISPR em todo o genoma identificam sensibilidade e resistência ao inibidor de PARP no câncer de próstata (2023, 4 de abril) recuperado em 5 de abril de 2023 em https://medicalxpress.com/news/2023-04-genome-wide-crispr-screens-parp- inibidor.html
Este documento está sujeito a direitos autorais. Além de qualquer negociação justa para fins de estudo ou pesquisa privada, nenhuma parte pode ser reproduzida sem a permissão por escrito. O conteúdo é fornecido apenas para fins informativos.