Cientistas descobrem os principais ‘culpados’ em estudo sobre câncer de pulmão

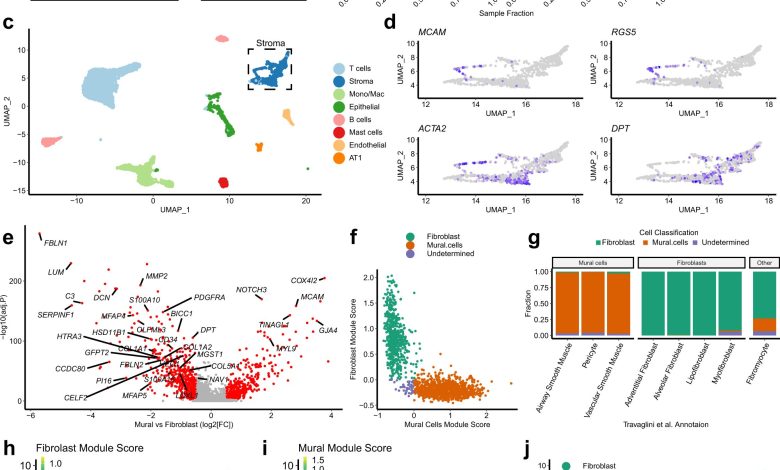
Identificação de fibroblastos por meio de análise de sequenciamento de RNA de célula única de homogenatos de tecidos inteiros derivados de amostras de tumor humano NSCLC. a Esquema ilustrando a metodologia de processamento e análise de amostra usada para gerar o conjunto de dados de drop-seq pulmonar alvo (TLDS), composto por amostras de controle humano (n = 6) e NSCLC (n = 12). A Figura foi parcialmente gerada usando a Servier Medical Art, fornecida pela Servier, licenciada sob uma licença Creative Commons Attribution 3.0 unported. b Barplots mostrando dados demográficos chave do conjunto de dados TLDS, mais detalhes fornecidos em Dados Suplementares 1. c. Visualização 2D (redução de dimensionalidade UMAP) de dados agrupados de dados scRNA-seq de tecido inteiro de todas as amostras, destacando diferentes tipos de células. Uma análise mais detalhada é mostrada na Fig. 1a-d suplementar. Células alveolares AT1 tipo 1, monócitos Mono/Mac e macrófagos. d Gráficos de recursos mostrando a expressão de marcadores canônicos para células murais (MCAM e RGS5), células murais e miofibroblastos (ACTA2) e fibroblastos (DPT), no agrupamento de células estromais (subconjunto do painel c). e Gráfico Volcano mostrando genes expressos diferencialmente (Bonferroni adj.P 0.5, mostrado em vermelho) entre células murais e fibroblastos de um atlas de células pulmonares humanas (HLCA) publicado recentemente. Os genes incluídos nas assinaturas genéticas de consenso identificadas para células murais ou fibroblastos são marcados. f Gráfico de dispersão mostrando a classificação de fibroblastos e células murais com base em assinaturas gênicas de consenso (definidas em e), aplicadas a células estromais do conjunto de dados HLCA. g Barplots mostrando os resultados dos resultados da classificação da assinatura do gene em comparação com a anotação do tipo de célula da publicação HLCA original. h Gráfico de recursos mostrando a expressão da assinatura do gene marcador de fibroblasto de consenso no conjunto de dados TLDS. i Gráfico de recursos mostrando a expressão da assinatura do gene do marcador mural de consenso no conjunto de dados TLDS. j Gráfico UMAP mostrando o resultado da demarcação de fibroblastos e células murais no conjunto de dados TLDS usando a abordagem de assinatura de gene de consenso. Crédito: Natureza Comunicações (2023). DOI: 10.1038/s41467-023-35832-6
O câncer de pulmão é a maior causa de morte por câncer no Reino Unido, mas um estudo realizado por uma equipe de cientistas da Universidade de Southampton descobriu uma nova maneira de identificar pacientes com duas vezes mais chances de morrer da doença.
Os pesquisadores do Centro de Imunologia do Câncer da Universidade usaram técnicas de ponta para aprender sobre um tipo particular de célula que envolve os tumores – chamada de fibroblasto– com mais detalhes do que nunca. Esses são células saudáveis que apóiam a cicatrização de feridas, mas podem ser sequestradas pelo câncer para ajudar os tumores a crescer e se espalhar.
Usando equipamentos criados por engenheiros universitários, juntamente com a tecnologia mais recente, eles conseguiram identificar três tipos diferentes de fibroblastos pela primeira vez – um miofibroblasto “sequestrado” e dois fibroblastos “normais”.
A pesquisa mostra que pacientes com alta proporção de miofibroblastos têm o dobro do risco de morrer de câncer de pulmão dentro de quatro anos em comparação com pacientes com menos miofibroblastos em seus tumores. A descoberta foi publicada na revista Natureza Comunicações.
O Dr. Chris Hanley, que co-liderou a pesquisa, disse: “Esses tipos de fibroblastos indicam se um paciente vai sobreviver por um período de tempo mais longo ou mais curto, então essa informação pode potencialmente ser usada para fornecer um prognóstico mais preciso.
“Também acreditamos que os pacientes que têm um alto número de miofibroblastos em seus tumores podem se beneficiar de novos tratamentos que podem atingir essa célula específica, o que acabaria por melhorar as taxas de sobrevivência”.
A equipe analisou 10.000 fibroblastos de 100 pacientes, decompondo-os um a um usando uma técnica chamada sequenciamento de célula única.
Usando uma máquina construída por engenheiros da Universidade de Southampton, os cientistas conseguiram capturar indivíduos células em uma gota, antes de estourar a célula para capturar as diferentes moléculas. Isso lhes permitiu contar quantas moléculas diferentes existem em uma determinada célula, dando-lhe uma impressão digital.
Esta técnica revelou os três tipos de fibroblastos, cada um com sua própria impressão molecular e função. Essas impressões digitais foram usadas para rastrear as origens dos fibroblastos e mostraram que apenas os miofibroblastos estavam diretamente ligados ao aumento do risco de morrer com câncer de pulmão.
Dr. Chris Hanley disse: “O que não estava claro até agora é se todos os fibroblastos são úteis para o câncer, mas este trabalho mostra que os miofibroblastos são os culpados mais perigosos. Se pudermos eliminar os miofibroblastos, teremos uma chance melhor de eliminar câncer de pulmão”.
Este é o maior estudo já realizado em fibroblastos em pacientes com câncer de pulmão, levando seis anos para ser concluído. Foi ideia do Dr. Chris Hanley e do Professor Gareth Thomas muito antes do Avanços tecnológicos estavam disponíveis para eles realizarem suas pesquisas.
O professor Thomas, colíder, disse: “Começamos essa ideia do zero quando esta era uma tecnologia muito nova e foi um longo caminho e uma quantidade incrível de trabalho para chegar a esse ponto. muitas oportunidades para projetos futuros e temos uma ideia muito mais clara de onde estamos indo por causa disso.
“O próximo passo é desenvolver formas de direcionar os fibroblastos ‘ruins’ para tornar os tratamentos mais eficazes em pacientes com câncer de pulmão de alto risco”.
O Dr. Iain Foulkes, diretor executivo de pesquisa e inovação da Cancer Research UK, disse: “Para vencer o câncer mais cedo, precisamos entendê-lo em detalhes. Graças a pesquisas pioneiras como esta, estamos nos aproximando do dia em que todos os pacientes poderão recebem tratamento adaptado às características de seu tumor.
“A tecnologia disponível para os pesquisadores de câncer hoje nos dá uma visão sem precedentes sobre como o câncer começa e cresce. Esperamos que esta pesquisa se torne a base de novos tratamentos para Câncer na clínica no futuro.”
Mais Informações:
Christopher J. Hanley et al, análise de célula única revela subpopulações de fibroblastos de prognóstico ligadas a subtipos moleculares e imunológicos de câncer de pulmão, Natureza Comunicações (2023). DOI: 10.1038/s41467-023-35832-6
Fornecido por
Universidade de Southampton
Citação: Cientistas descobrem os principais ‘culpados’ no principal estudo sobre câncer de pulmão (2023, 1º de fevereiro) recuperado em 1º de fevereiro de 2023 em https://medicalxpress.com/news/2023-02-scientists-key-culprits-major-lung.html
Este documento está sujeito a direitos autorais. Além de qualquer negociação justa para fins de estudo ou pesquisa privada, nenhuma parte pode ser reproduzida sem a permissão por escrito. O conteúdo é fornecido apenas para fins informativos.

