O sequenciamento multimodal alcança resultados de alta qualidade a partir de pequenos volumes de espécimes tumorais congelados

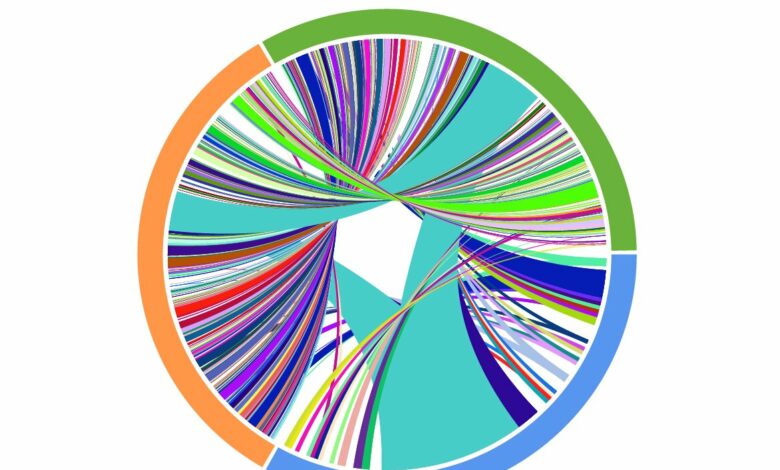
Um gráfico de Circos representando os resultados do sequenciamento de receptores de células T correspondentes coletados em um paciente submetido a terapia anti-PD1. As cores indicam a hora da coleta (azul: pré-tratamento, laranja: no tratamento, verde: no-mais tarde) e as conexões indicam a sobreposição de TCRs idênticos. Esses resultados demonstram uma notável diversificação de clonótipos de células T ao longo do tempo. Crédito: Yiping Wang/CUIMC
Pesquisadores da Columbia Engineering e do Columbia University Irving Medical Center inventaram um novo método de sequenciamento de RNA que obtém resultados de alta qualidade a partir de pequenos volumes de amostras de tumores congelados. Eles demonstraram o sucesso de sua técnica em dois estudos clínicos que traçaram o perfil de dezenas de amostras de tumores, tanto as arquivadas quanto as coletadas recentemente, para entender como elas respondem à terapia antitumoral. O artigo foi publicado em 9 de janeiro de 2023, por Genética da Natureza.
Usando o sequenciamento de RNA para medir expressão genetica na resolução de células individuais tem sido uma das ferramentas mais transformadoras para estudar tecidos de câncer na última década. Ao examinar o RNA de células individuais, os pesquisadores podem entender melhor a diversidade de células dentro de um tumor, bem como a forma como essas células tumorais crescem e interagem com as células imunes. Esses são fatores importantes para entender as características da progressão do câncer e a resistência do câncer à terapia – ambos essenciais para o desenvolvimento de novos tratamentos contra o câncer.
Uma grande barreira para a adoção generalizada de sequenciamento de RNA de célula única em fluxos de trabalho clínicos tradicionais tem sido o volume de tecido fresco necessário – substancialmente mais do que o que é rotineiramente coletado para fins clínicos. E a necessidade de tecido fresco significa que as amostras devem ser analisadas imediatamente após a coleta. Esses requisitos limitaram significativamente as análises científicas que podem ser feitas em amostras de pacientes.
Nova técnica de sucesso
Focada em superar essas barreiras, a equipe de Columbia criou um novo método de sequenciamento que forneceu os melhores resultados a partir de uma pequena quantidade de espécimes de tumores congelados. Geralmente coletados durante ensaios clínicos e armazenados em biobancos, esses espécimes podem incluir tecidos de cânceres raros e pacientes com histórias únicas ou fatores de risco. A capacidade da nova técnica de sequenciar esses tipos de espécimes aumenta muito o número e a variedade de amostras de tumores disponíveis para análise científica.
“Estamos muito entusiasmados com este trabalho e seu potencial”, disse Elham Azizi, professor assistente de engenharia biomédica e professor assistente Herbert e Florence Irving de pesquisa de dados sobre câncer no Irving Institute for Cancer Dynamics, que co-supervisionou este estudo com Benjamin Izar. , professor assistente de medicina no Herbert Irving Comprehensive Cancer Center.
“A capacidade de trabalhar com amostras congeladas abre as portas para colaborações multi-institucionais que impulsionarão a descoberta de biomarcadores e alvos de drogas. Também nos dá a oportunidade de aplicar técnicas computacionais de ponta na análise e integração dos dados clínicos desbloqueados “, explicou Azizi.
Izar acrescentou: “E como nosso método requer apenas uma quantidade mínima de tecido, o restante da amostra pode ser usado para estudos adicionais. Isso realmente é vantajoso para pesquisadores, médicos e, mais importante, para nossos pacientes”.
Os resultados do estudo ampliam a compreensão da progressão do câncer
O novo estudo liderado por Yiping Wang, Joy Fan e Johannes Melms, estagiários nos Laboratórios Izar e Azizi, gerou resultados de sequenciamento de RNA de célula única, sequenciamento de receptor de célula T de célula única, sequenciamento completo do genomae sequenciamento espacial de RNA (um método inovador de sequenciamento de RNA que preserva a arquitetura do tumor in situ) – todos realizados nas mesmas amostras.
Com a capacidade de conectar várias modalidades de dados sobre o câncer, os pesquisadores forneceram uma visão abrangente das alterações genéticas, funções celulares, dinâmica das células imunológicas e localização espacial das células no contexto do tecido do paciente. Essas melhorias ampliaram significativamente sua compreensão da progressão do câncer e dos mecanismos de resistência.
A equipe agora está aplicando suas novas técnicas experimentais e computacionais para analisar coortes clínicas maiores. Os dados permitem que eles estudem melhor a progressão da doença e examinem o impacto das terapias em ensaios clínicos em melanoma, bem como em outros tipos de câncer.
ECHIDNA, uma nova ferramenta computacional
Paralelamente, os pesquisadores também estão desenvolvendo uma ferramenta computacional inovadora para análise integrativa sistemática de dados do genoma inteiro e sequenciamento de RNA de célula única, chamada ECHIDNA. Eles esperam que esse algoritmo de aprendizado de máquina seja a chave para entender a relação entre alterações genéticas (genótipo) e função celular (fenótipo) em células tumorais e o aplicarão a uma coorte maior de pacientes com melanoma para caracterizar diversos mecanismos de resistência ao tratamento. O trabalho futuro também incluirá a construção de novos métodos computacionais para integrar esses dados dentro do contexto espacial da fatia de histologia, a fim de caracterizar a dinâmica espacial das interações tumor-células imunes.
Izar observou: “Além de compreender o tecido canceroso, nosso método também permite estudar a resposta do tecido e a imunologia do tecido em outras doenças. No passado, usamos uma versão anterior do método apresentado aqui para estudar a resposta do tecido à infecção letal por COVID-19 em vários órgãos. Isso demonstra um exemplo de desenvolvimentos originados em Pesquisa sobre câncer que podem impulsionar a pesquisa em ciências da vida em geral.”
Mais Informações:
Yiping Wang et al, Sequenciamento multimodal de célula única e genoma completo de pequenos espécimes clínicos congelados, Genética da Natureza (2023). DOI: 10.1038/s41588-022-01268-9
Citação: O sequenciamento multimodal alcança resultados de alta qualidade a partir de pequenos volumes de amostras de tumores congelados (2023, 18 de janeiro) recuperados em 18 de janeiro de 2023 em https://medicalxpress.com/news/2023-01-multimodal-sequencing-high-quality-results- small.html
Este documento está sujeito a direitos autorais. Além de qualquer negociação justa para fins de estudo ou pesquisa privada, nenhuma parte pode ser reproduzida sem a permissão por escrito. O conteúdo é fornecido apenas para fins informativos.