Detecção orientada a dados de variantes SARS-CoV-2 meses de antecedência

Análise contínua de genoma viral fornecida pela plataforma de cobertura (SarScoverage.org). Crédito: Comunicações da natureza (2025). Doi: 10.1038/s41467-025-60231-4
Desde o início da pandemia SARS-CoV-2, várias variantes do vírus se transformaram em variantes de preocupação (COV), conforme classificado pela Organização Mundial da Saúde (OMS). Os COV são variantes de vírus previstas ou conhecidas por causar grandes ondas de infecções devido a suas características fenotípicas alteradas e com o risco de alterar a gravidade da doença, reduzir a eficácia da vacina ou levar ao aumento da carga dos sistemas de saúde.
A plataforma da Web de cobertura para vigilância genômica do vírus SARS-CoV-2 permite uma identificação computacional rápida e caracterização de possíveis variantes de interesse (PVOIs), com um tempo de entrega de quase três meses antes da designação da OMS como um VOC ou categorias de variantes relacionadas e prever previsto sua capacidade de escapar da imunidade existente adquirida por vacinas anteriores ou de vacinas anteriores.
Pesquisadores liderados por Alice McHardy demonstraram isso com sucesso em uma análise abrangente publicada em Comunicações da natureza. A detecção precoce de COV é particularmente importante para o desenvolvimento da vacina, a fim de garantir a proteção da vacina contra novas variantes de vírus.
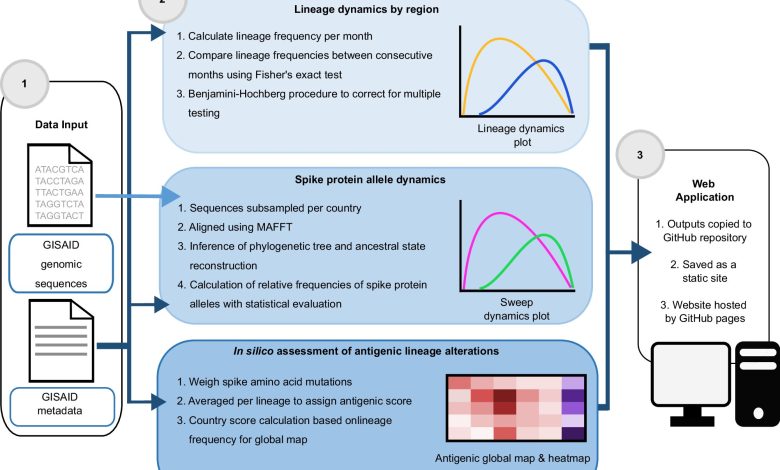
“Desenvolvemos um novo método de análise para cobertura que deve ajudar a tornar mais visíveis as alterações antigênicas nas variantes de vírus”, explica McHardy. Especificamente, é usada uma matriz baseada em observações do desenvolvimento a longo prazo de certos vírus influenza (influenza A H3N2). Essa matriz vincula mudanças importantes no material genético do vírus às suas propriedades.
Os pesquisadores estão analisando particularmente as mudanças em uma proteína específica do vírus, conhecida como proteína de pico. Essa proteína desempenha um papel importante porque permite que o vírus se prenda às células humanas e porque é o principal alvo para vacinas e terapias.
O sistema de cobertura obtém os dados relevantes do banco de dados do genoma do vírus Gisaid, que é uma iniciativa de compartilhamento de dados que promove a rápida troca de dados sobre patógenos prioritários como influenza, HCOV-19, RSV, HMPXV, SARS-COV-19 e arbovírus, como Chikungunya, Dengue, Zikungunya, Dengue, Zikungunya, Dengue, Zikungunya, Dengue, Zikungunya, Dengue, Zikungunya, Sars-Cov-19 e Arbovírus. Em março de 2024, o Gisiad tinha mais de 16,5 milhões de sequências SARS-CoV-2 disponíveis.
A cobertura analisa os dados do genoma SARS-COV-2 por país de origem para dinâmica de tensão e alterações antigênicas. Um método estatístico é usado para determinar quais cepas virais mudaram significativamente sua capacidade de fuga imune. Isso envolve a comparação das alterações de aminoácidos que ocorrem na proteína de pico de cepas virais de um determinado mês. As cepas que claramente se destacam – ou seja, aquelas que mostram mudanças significativamente maiores que a média – são selecionadas como significativamente alteradas.
As cepas virais conspícuas que se prevêem se espalharem mais rapidamente ou serem significativamente alteradas em termos de imunidade são exibidas na plataforma de cobertura em gráficos especiais (“mapas de calor”) para que os usuários possam ver rapidamente quando e onde grandes mudanças no vírus estão ocorrendo.
Para testar a confiabilidade do novo método de análise, os pesquisadores examinaram dados de sequência do genoma de cepas de vírus já conhecidas por serem COV, incluindo a variante omicron de SARS-CoV-2. O grupo de trabalho descobriu que o novo método permitia que as linhas de vírus fossem identificadas retrospectivamente como VOCs até três meses antes da designação da OMS.
“Foi interessante ver que as variantes de vírus que também foram oficialmente classificadas como importantes pela OMS mostraram valores significativamente mais altos em nossas análises do que outras variantes menos notadas”, explica McHardy. Os números subiram em uma ordem clara: primeiro para variantes que estão sendo monitoradas apenas (variantes sob monitoramento ou Vums), depois para variantes de interesse (VOIs) e, finalmente, mais fortemente, para as variantes VOC, que são consideradas particularmente preocupantes.
“No geral, esses resultados ressaltam a capacidade de nosso método de prever efetivamente o surgimento de variantes SARS-CoV-2 relevantes para a saúde com uma vantagem de crescimento-bem antes de atingirem sua frequência máxima ou são formalmente identificadas pela OMS como a respeito”, resume o bioinformático. “Isso pode fornecer um tempo valioso para iniciar uma análise aprofundada necessária para ajustes de vacinas ou tomar medidas direcionadas para proteger grupos vulneráveis, por exemplo”.
Mais informações:
Katrina Norwood et al, na vigilância genômica de silico por cobertura prevê e caracteriza variantes de interesse SARS-CoV-2, Comunicações da natureza (2025). Doi: 10.1038/s41467-025-60231-4
Fornecido pelo Centro Alemão de Pesquisa de Infecção
Citação: Detecção orientada a dados de variantes SARS-CoV-2 meses de antecedência (2025, 18 de julho) recuperadas em 20 de julho de 2025 de https://medicalxpress.com/news/2025-07-driven-cars-cov-variants-months.html
Este documento está sujeito a direitos autorais. Além de qualquer negociação justa para fins de estudo ou pesquisa particular, nenhuma parte pode ser reproduzida sem a permissão por escrito. O conteúdo é fornecido apenas para fins de informação.